
抗体由两条相同的重链和两条相同的轻链组成,是一种呈现Y形结构的糖蛋白(~150kDa)。重链和轻链的可变区负责与抗原结合,而恒定区则与免疫系统的其他成分作用产生相应效应功能。重链轻链可变区均包含三个超变环,称为互补决定区(CDR1、CDR2和CDR3)。CDR区被称为框架区(FR1、FR2、FR3和FR4)的结构保守区域间隔开,这些区域形成可变区“核心”β片结构,超变环展示在其表面。CDR序列的长度和组成变化很大,尤其是CDR3。在抗体工程过程中通常需要精确鉴定对抗体与其靶抗原的相互作用和/或亲和力有影响的残基,需要准确鉴定出CDR区。本文介绍了现阶段常用的抗体编号方案,并比较解释了由此方案定义CDR的优势与不足。
随着抗体测序和晶体分析技术的不断发展,各种序列和结构数据库数量显著增加,使得人类和动物免疫球蛋白的可变区的比较成为可能。1970年,Kabat和Wu比对了77个Bence-Jones蛋白和免疫球蛋白轻链序列,研究抗体可变区连续位置氨基酸组成的统计学上的变异性。他们将“变异性”定义为给定位置上不同氨基酸的数量除以该位置上出现最多氨基酸的频率。该分析揭示了轻链可变区中的三个高变区,并证明了高度保守的残基的存在。例如在抗体结构域内核心形成二硫键的两个半胱氨酸和位于CDRL1后的色氨酸残基。同样,在重链可变区也发现了三个相应的高变区。Kabat等假设这些高变区会聚集在折叠结构域的一侧,形成负责特异性抗原识别的表面,并将这些高变区称为互补决定区(CDR1、CDR2和CDR3)。这一假设后来得到证实,并进一步研究区分这些CDR中抗原结合或构象的重要残基。
1979年,Kabat等首次提出了抗体可变区的标准化编号方案。在他们编制的《免疫学意义蛋白质序列》中,对抗体重链和轻链(λ,κ)的可变区以及T细胞受体(α,β,γ,δ)的可变区的氨基酸序列进行了比对和编号。他们观察到,分析的序列表现出可变的长度,并且只能在精确的位置产生缺失和插入。有趣的是,插入点位于CDR内部(除CDRL2),以及位于框架区内的部分位置。在这套编号方案中,这些插入残基可以被识别并用字母注释(例如,27a、27b……)。值得注意的是,所有λ轻链中都不存在残基L10,因为λ和κ链由位于不同染色体上的两个不同基因编码。
尽管Kabat编号方案被广泛采用,但它有一些局限性:首先,该方案建立在有限数量具有常见序列长度的抗体的序列比对之上。因此,在CDR或框架区中具有非常规插入或缺失的序列不包括在内。其次,Kabat编号方案与抗体的3D结构不能很好地匹配。事实上,Kabat定义的高变区与抗原结合环结构并不完全匹配。CDRL1(L27)和CDRH1(H35)中定义的插入位点与其在结构中的相应位置不匹配,换言之,CDRL1和CDRH1中晶体结构中相应的残基(拓扑对齐)与Kabat编号方案中编号不同。
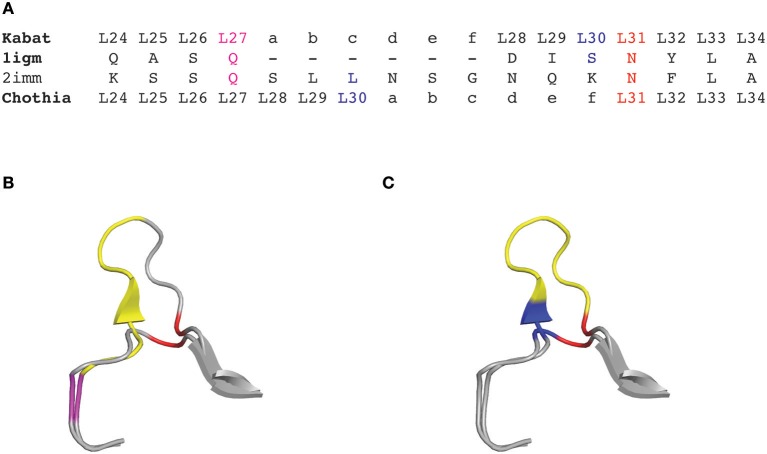
图1 轻链可变区1igm和2imm根据Kabat和Chothia编号方案CDRL1氨基酸插入位置
(A)Kabat/Chothia编号方案1igm和2imm CDRL1的氨基酸序列
(B)根据Kabat编号方案(C)根据Chothia编号方案1igm和2imm的CDRL1环的飘带图
1987年,Chothia等为抗体可变区引入了基于结构的编号方案。他们对抗体可变区的晶体结构进行了比对,定义了形成CDR的环结构,并校正了CDRL1和CDRH1插入点的位置编号,以便更好地拟合其拓扑位置(图1)。
根据抗体结构的比对,Chothia编号方案将氨基酸插入点从L27移动到L30,H35移动到H31。值得一提的是,Chothia CDR的定义与抗体环结构的对应更好。Chothia鉴定的CDRH3环结构与Kabat高变区匹配性较好,相比之下其他环比Kabat定义的超变序列短(CDRH1除外)。根据Kabat编号方案高变氨基酸基础上定义的CDR和基于Chothia编号方案的环拓扑结构的CDR对于部分CDR有位置移动和/或环长度偏离(图2)。

图2 1kiq结构根据Kabat(A、C)和Chothia(B、D)编号方案在CDR定义上的差异
A、B为轻链,C、D为重链可变区3D结构,框架区用灰色表示,CDR用红色表示
Chothia编号方案的主要优点在于不同抗体拓扑对齐残基位于相同的位置编号,并且Chothia CDR定义在大多数抗体序列中对应于抗原结合环。然而,与Kabat或IMGT编号方案相比,这种编号方案的使用有限,可能产生混淆。此外,Chothia等后来发表的一项研究将CDRL1中的插入点从L30更改为L31,但大型数据库研究抗原结合环构象时还是沿用最初的L30位置。轻链也是如此,他们将λ链L93插入点移动到L95。与Kabat编号方案类似,Chothia编号方案的一个重要限制是忽略了非常规长度的序列。也同样可以通过定义新的插入点来优化该系统。
Martin等2008年的研究重点关注非常规长度的不同框架区的结构比对。他们指出了大多数序列和结构中不存在的残基,并将其定义为缺失位置。通过分析序列和结构,他们提出重链FR3内的插入点应从H82改到H72。此外,通过与CDRH2的类比,将CDRL2的插入点位置修改到L52。
他们在Chothia编号系统的基础上提出了一种新的编号方案,并开发了新的编号软件ABnum(http://www.bioinf.org.uk/abs/abnum/)。该软件使用了更大的Abysis数据库,集成了来自Kabat、IMGT和PDB数据库的序列。ABnum在Chothia和Kabat编号方案中H6定义了一个新的插入/缺失位置,而Martin编号方案这个位置在H8。
Martin编号方案应被视为Chothia编号的新版本。通过分析大型数据库上的序列和结构,可以校正插入位置,定义新的插入位置并突出显示缺失的位置,更好地拟合残基的拓扑位置。
一些研究中使用的另一种有趣的编号方法是Gelfand等描述的编号方法。这种编号系统选用了相对复杂的命名方法。可变区序列被分成21个片段,称为“word”,这些“word”中的每一个都与二级结构元素(strand或loop)匹配。strand区由按顺序排列的字母定义(例如,A、B、C),loop区由对应于相邻strand的两个字母定义(例如,AB、BC…..)。该定义有两个例外:可变区的三个N端残基(称为OA)和连接B和C strand的loop环,因为它存在一个“双跨桥”构象(图3),这个loop分为两个word,分别为BC和CB。该编号系统不包括插入或缺失点,但可以对比对序列之间的二级结构进行精确比较。同样值得注意的是,Gelfand对loop的定义与Chothia的并不完全对应。
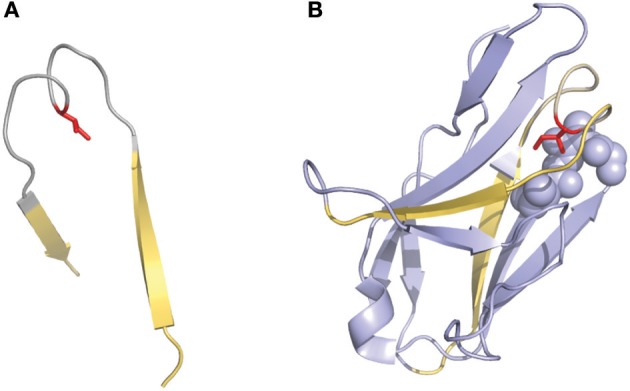
图3 2fb4抗体结构中存在的CDRL1的“双跨桥”构象
双跨桥用灰色表示,边界strand用黄色表示
(A)Ile侧链(红色)指向loop环结构内部(B)完整的结构域
1997年,Lefranc等为免疫球蛋白超家族的所有蛋白序列引入了一种新的标准化编号系统,包括来自抗体重链轻链和的可变区以及来自不同物种的T细胞受体链。该编号方案开始基于种系V基因的氨基酸序列比对,氨基酸序列和编号在CDR3开始的地方停止。后来,Lefranc将编号方案扩展到整个可变区,并开发了各种工具来分析全长序列。IMGT拥有自己的框架区(称为FR-IMGT)和CDR(称为CDR-IMGT)定义。
IMGT编号方案根据种系V序列比对从1到128连续计数残基。除了在位置111和112之间具有13个以上氨基酸的CDR3-IMGT,无其他插入位置。当特定序列中缺少残基时,则该编号为空。例如,在6个氨基酸长的CDR1-IMGT中,残基#27后跟残基#34(编号#28–#33无残基)。根据Kabat、Chothia和IMGT编号方案的比对示例如图4所示。
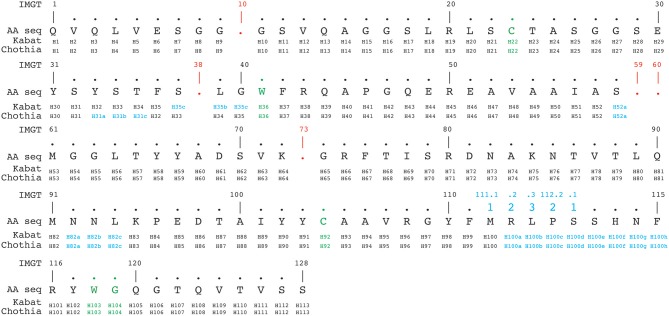
图4 根据Kabat、Chothia和IMGT编号方案对3dwt纳米抗体序列进行比对
IMGT是免疫遗传学和免疫信息学的重要参考。其定义,包括其氨基酸编号方案已得到广泛认可,并被世界卫生组织国际免疫学会联合会使用。这种编号方案的主要优点是基于来自包括整个免疫球蛋白超家族的完整参考基因数据库的序列比对。相应工具也不断开发,如氨基酸比对和编号可以通过IMGT/DomainGapAlign执行。该工具还能够通过识别编码可变区的相应VDJ基因来分析序列多态性。IMGT与另一个有趣的程序相结合,称为IMGT-“CollierdePerles”,可以一目了然地看到氨基酸2D位置,并且还可以轻松标注FR-IMGT和CDR-IMGT。
然而,由于氨基酸沿序列连续编号,IMGT编号方案无法直观地显示插入位置,即使对于最常见的插入位置也是如此。在Kabat和Chothia编号系统中,氨基酸插入点的位置很容易合并;将IMGT方案应用于具有新氨基酸插入的潜在序列更加困难。必须注意的是,IMGT将插入片段置于CDR的末尾,这与抗体结构无关。这个问题在后来的V-Quest软件中得到了纠正,该软件将插入片段放在CDR-IMGT的中间,这与实际结构数据更匹配。
Honegger编号方案以均一化格式对免疫球蛋白超家族的可变区进行编号。该系统基于免疫球蛋白可变区3D结构的结构对齐,覆盖观察到的长度变化。它允许定义结构保守的Cα位置,并推断出适当的框架区和CDR长度(图5)。
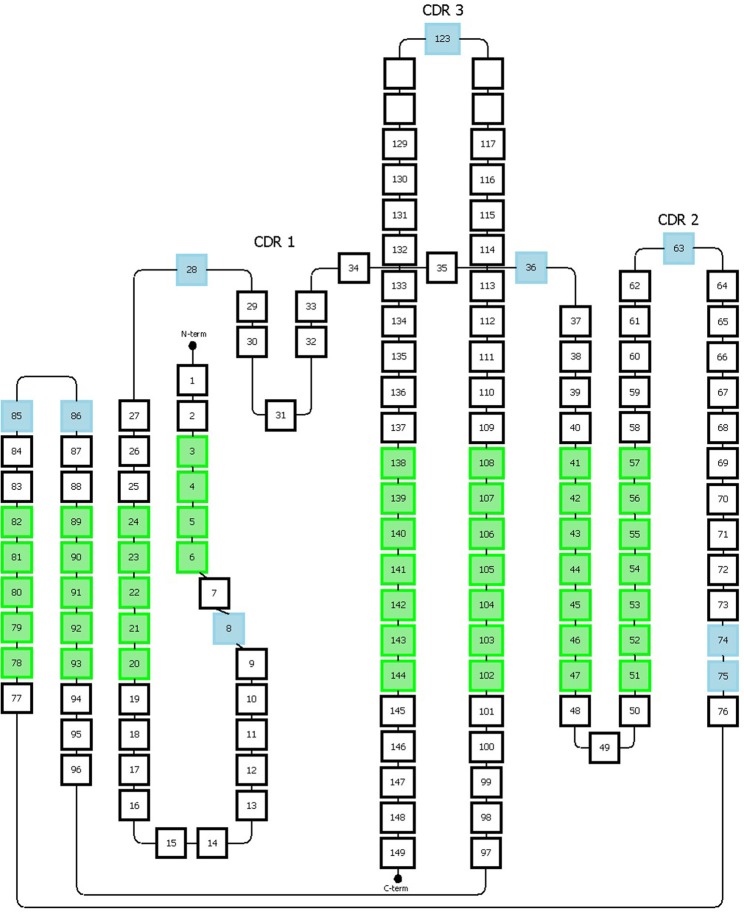
图5 Honneger编号方案
提供缺失位置的氨基酸用蓝色表示;绿色位置对应于结构保守的残基,其Cα位置用于结构叠加
Honegger编号方案还定义了保守残基(C23、W43、C106、G140)和特定位置的缺失(#27-28、#36、#63、#123)。CDR1具有“双跨桥”构象,该构象由位置#31处的保守疏水残基深入结构中产生,将loop环分为两个部分。Honegger方案描述了位于这两个部分上的两个缺失区域,一个位于第一部分#27和#28,另一个位于第二部分#36。该方案考虑了loop环两端存在的插入位点的可变性。此外,另外两个插入点分别位于位置#74和#75,以反映T细胞受体α表现出的CDR2环的较短C端分支。额外的缺失位置在CDR2和CDR3环的中间。通过进一步的结构分析,他们提出了将Vκ链插入缺失从最初L10移动到L8。
Honegger编号系统的主要优点在于它基于结构比对,可以更好地匹配抗体3D结构特征。此外,Honegger在CDR1和CDR2中包含两个缺失,非常适合对免疫球蛋白超家族中的所有蛋白进行编号。然而,与IMGT方案类似,Honegger会跳过部分顺序编号,这在分析序列编号时可能会令人困惑。这种编号方案也不太灵活,无法适用于具有新插入或较大插入的免疫球蛋白。结构最保守位置的分析仅来自28个不同的结构,通过针对特定类型的免疫球蛋白(例如抗体)的可变区调整方案,可能可以更好地定义框架区。
| Loop | Kabat | AbM | Chothia | IMGT |
|---|---|---|---|---|
| L1 | L24-L34 | L24-L34 | L26-L32 | L27-L32 |
| L2 | L50-L56 | L50-L56 | L50-L52 | L50-L51 |
| L3 | L89-L97 | L89-L97 | L91-L96 | L89-L97 |
| H1(Kabat Numbering) | H31-H35B | H26-H35B | H26-H32..34 | H26-H35B |
| H1(Chothia/Martin Numbering) | H31-H35 | H26-H35 | H26-H32 | H26-H33 |
| H2 | H50-H65 | H50-H58 | H52-H56 | H51-H56 |
| H3 | H95-H102 | H95-H102 | H96-H101 | H93-H102 |
参考文献
[1]Dondelinger M, Filée P, Sauvage E, et al Understanding the Significance and Implications of Antibody Numbering and Antigen-Binding Surface/Residue Definition. Front Immunol. 2018 Oct 16;9:2278.
[2]Martin, A.C.R. Protein Sequence and Structure Analysis of Antibody Variable Domains. In: Kontermann, R., Dübel, S. (eds) Antibody Engineering. Springer Lab Manuals. Springer, Berlin, Heidelberg,2001.

南京德泰生物工程有限公司 Nanjing Detai Bioengineering Co.,Ltd. ©2025 All Rights Reserved