
免疫球蛋白的特异性由可变区高变区序列和长度决定,这些区域产生与抗原互补界面。先前研究比较了已知的高变区序列,发现在轻链的13个位点和重链的7个位点的残基是保守的。研究者认为,影响抗体结构的是这些保守残基位点,而不是整个高变区。这些残基在抗体中有固定的位置,可以用于结合位点的模型构建,以限制残基变化的位点的构象和位置。同时,对Fab和Bence-Jones蛋白结构分析的结果表明,长度相同但序列不同的高变区具有相同的主链构象。
基于以上认识,Chothia等通过对已知免疫球蛋白原子结构的分析,确定了已知结构共同的β-片层框架,确定了相对较少的残基,这些残基通过堆积、氢键或构成不寻常的φ、ψ或ω构象的能力,是高变区中观察到的主链构象的主要原因。这些残基一般存在于高变区和β-片层保守框架区。
如果上述残基存在于其他免疫球蛋白序列中,那么它们的高变区应与具有相同特征的已知结构高变区具有相同的构象,这些构象被称为“典型结构(canonical structures)”。如果在不影响特定规范结构构象的位点发生的序列变异,将会改变其呈现给抗原的表面。
研究基于Kabat等已经发表的一系列免疫球蛋白序列:对于轻链的可变区VL,包含大约200个完整序列和400个部分序列;对于重链的可变区VH,包含大约130个完整和200个部分序列。使用Kabat编号方案对这些序列进行编号,在少数情况下,某些高变区由结构叠加给出了不同于序列比对所建议的结果。
在表1中列出了已知结构的免疫球蛋白,其原子坐标可从蛋白质数据库(Protein Data Bank)获得,并提供了晶体图分析的参考。
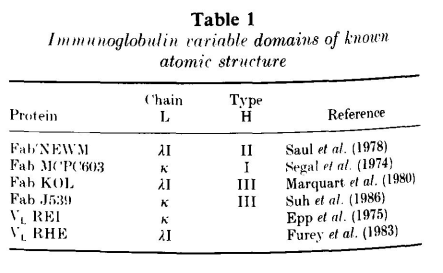
VL和VH结构域具有同源的结构。每个结构域都包含两个大的β-片层,以主链间1 Å的距离相互包裹(1 Å=0.1 nm),以-30°的角度倾斜(图1)。每个结构域的β-片层由一个保守的二硫键连接。抗体结合位点由6个高变区形成,这些区域连接β-片层的β链。其中两个高变区是由不同β-片层的β链相连,另外四个是发夹弯——连接同一折叠中相邻两条β链的多肽(图2)。中小型发夹弯的构象主要取决于转角的长度和序列。表2总结了关于发夹弯构象的研究结果。
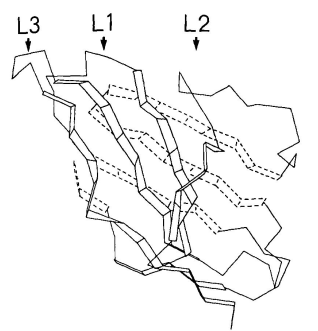
图1 免疫球蛋白可变区结构。
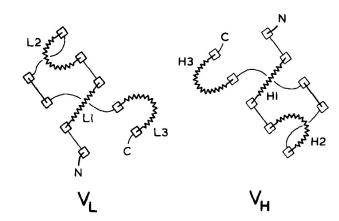
图2 免疫球蛋白高变区结合位点排列
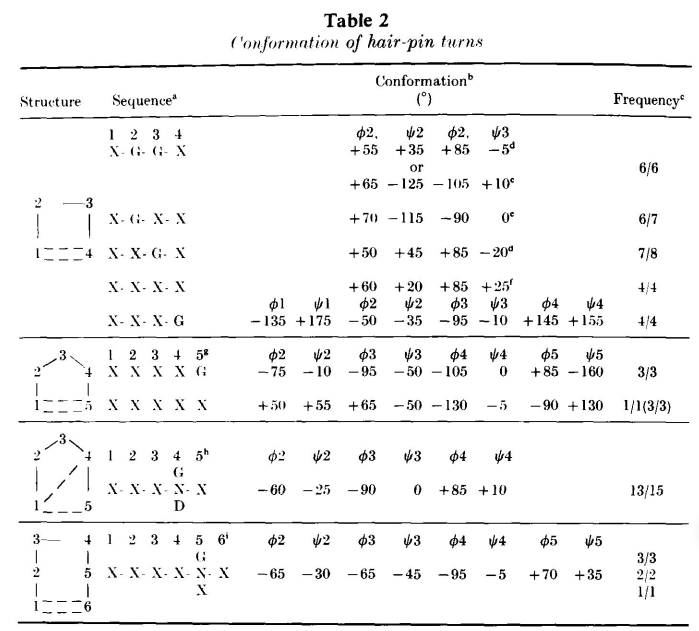
不同免疫球蛋白分子的β-片层框架区非常相似。可变区框架区的结构相似性是由于形成域内部的残基趋向保守,以及内部残基总体量保守。通过对已知结构免疫球蛋白的主链原子进行最佳叠加比对(表1),计算同源残基原子位置的差异可以确定不同结构的相似性。图3给出了VL和VH β-片层框架的方案。β-片层框架包括VL残基4~6、9~13、19~25、33~49、53~55、61~76、84~90、97~107和VH残基3~12、17~25、33~52、56~60、68~82、88~95和102~112。
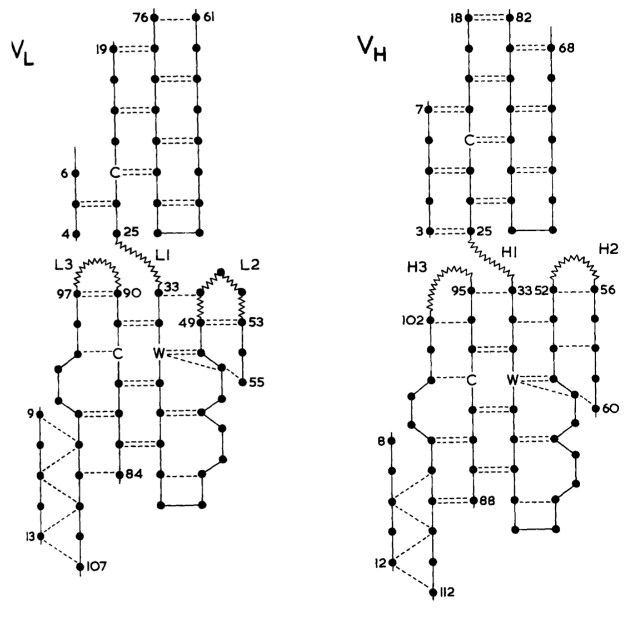
图3 β-片层框架折叠方案
框架的三级结构的主要决定因素是域内和域间的残基。表4列出了通常掩埋在VL和VH域中以及域间界面中的残基及它们的可及表面积。这些残基通常表现得较为保守。
VL序列中有两个位点的保守残基性质取决于链的类型。Vλ 71位和90位残基通常分别为Ala和Ser/Ala;而Vκ相应残基通常为Tyr/Phe和Gln/Asn。这些残基与高变区相连,并对环构象产生一定的影响。
框架结构的保守性扩展到紧邻高变区的残基。若对一对免疫球蛋白分子的框架区进行比对,这些残基位置的差异在大多数情况下小于1 Å(表5)。相反,在与框架区相邻的高变区残基,位置可以相差3 Å或更多。
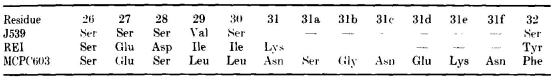
6个主链构象不同的环状结构是抗体结合位点的一部分,分别由VL结构域L1、L2、L3于26~32、50~52和91~96位点和VH结构域H1、H2和H3的26~32、53~55和96~101位点组成,与Kabat等根据序列变异性定义的互补决定区有些不同:VL的24~34、50~56和89~97,VH的残基31~35、50~65和95~102。
在已知的VL结构中,L1区残基26至32的构象展示了轻链的类别特征。在Vλ结构域中,构象呈螺旋状,而在Vκ结构域中,构象呈延伸状。这些构象差异是L1区和框架的序列差异造成的。
RHE和KOL的L1区包含9个残基,分别为26至30、30a、30b、31至32,NEWM多一个残基。RHE和KOL的L1区具有相同的构象:它们的主链原子位置仅有0.28 Å的均方根(r.m.s.)差异。将NEWM的L1区与KOL和RHE的L1区叠加显示,额外的残基插入在残基30b和31之间,对该区域其余部分的构象影响很小;将NEWM中26至30b和31至32的主链原子与KOL和RHE中26至32的主链原子叠加,分别有0.96 Å和1.25 Å的均方根差异。通过结构叠加得出的KOL、RHE和NEWM Vλ L1区的序列比对如下:

在这三种结构中,残基26的羰基和残基29的酰胺之间形成氢键Ⅰ型转角。残基27至30b形成一个不规则的螺旋(图4),该螺旋位于β-片层核心的顶部。残基30的侧链深入核心,占据了残基25、33和71之间的空腔。在观察到的结构中,决定L1构象的主要因素是残基25、30、33和71的堆积。Vλ RHE、KOL和NEWM在这些位点具有相同的残基:Gly25、Ile30、Val33和Ala71。(L1残基Asp29或Asn29因与L3接触而被掩埋)。
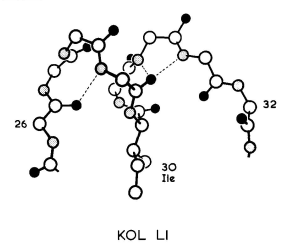
图4 KOL Vλ结构域L1区构象
根据Kabat等列出的33个L1区序列已知的人类Vλ结构域,其中Ⅰ、Ⅱ、Ⅴ和Ⅵ亚组中的21个序列的L1区长度与RHE、KOL或NEWM中的序列相同。其中18个序列保留了上述构象的关键残基,这意味着这18个L1区的构象与RHE、KOL或NEWM中的构象相同:

而亚群Ⅲ和Ⅳ中有13个L1区序列已知,长度比RHE和KOL以及其他Vλ亚群的序列更短,保守残基模式也大不相同。
根据Kabat等列出的29个L1区序列已知的小鼠Vλ结构域。这些L1区的长度与NEWM中的相同,它们的保守残基模式也与KOL/NEWM中的相似,但不完全相同:Ser25,Val30,Ala33,Ala71。
图5展示了三个已知的Vκ结构:J539、REI和MCPC603中L1区的构象。J539的L1有6个残基,REI有7个,MCPC603有13个。J539的L1区具有伸展构象(extended conformation)。REI中残基26至28具有伸展构象,残基29至32形成扭曲的Ⅱ型转角。
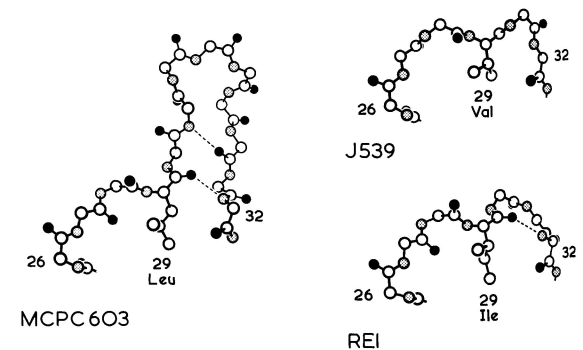
图5 Vκ结构域L1区构象
MCPC603中的另外6个残基都出现在这一转角区域。在这三种结构中,残基26至29和32的主链具有相同的构象。对J539、REI和MCPC603中这些残基进行叠加,得出的位置r.m.s.差异为0.47 Å至1.03 Å。结构叠加所得出的序列比对结果为:
在J539、REI和MCPC603中,残基26至29延伸至整个β-片层框架的顶部,其中一个残基29被掩埋在其中,残基29的主要接触点是残基2、25、33和71。残基29对框架内部的渗透不如残基30在Vλ结构域中的渗透大,Vκ结构域中存在的深腔被残基71的大侧链填满。在J539、REI和MCPC603中,参与L1堆积的残基(2、25、29、33和71)非常相似,分别是Ile、Ala/Ser、Val/Ile/Leu、Leu和Tyr/Phe。
MCPC603中的6个残基30至30f形成了一个发夹环,从结构域中延伸出来,没有良好有序的构象。
Kabat等曾指出,当时已知的Vκ序列L1区某些位置上的残基是保守的,并认为这些残基具有结构性作用。上述对Vκ结构的分析以及目前已知的大量序列中残基保守模式证实了残基25、29和33的结构作用。
根据Kabat等列出的65个人类Vκ序列和164个小鼠Vκ序列,有59个人类序列和148个小鼠序列在涉及L1区包装的位点上的残基与已知结构中的残基非常相似:

这些序列中L1区的残基数量各不相同:
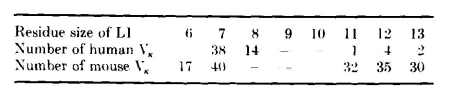
掩埋在L1区和框架之间位置的残基的保守性意味着,在绝大多数Vκ结构域中,残基26至29的构象与已知结构中的构象接近,其余残基如果数量少,则形成一个转角,如果数量多,则形成一个发夹环。
在已知结构中,L2区具有相同的构象。L2结构的相似性源于三残基转角的构象要求和L2包裹的框架区残基的保守。已知结构的L2区由三个残基组成,50至52:

这三个残基连接框架β-片层中两条相邻的链。残基49和53以氢键连接,L2区形成由三个残基组成的发夹转角。
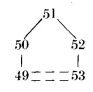
这五种结构L2区的构象非常相似:其主链原子位置的r.m.s.差值在0.1 Å到0.97 Å之间。构象间的唯一差异在于残基50和51之间的肽的方向。在MCPC603中,这种差异与50位的甘氨酸残基有关(图6)。
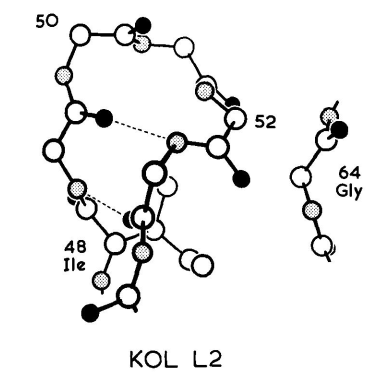
图6 KOL Vλ结构域L2区构象
L3区域(残基91至96)是两条相邻的β-片层链之间的连接区域。对该区域已知结构和序列的分析表明,绝大多数κ链拥有共同构象,且与λ链的构象截然不同。
NEWM Vλ L3区有6个残基,KOL和RHE有8个残基:

在所有三种Vλ结构中,残基91至92和95至96构成了β-片层框架结构的延伸,残基92和95之间存在主链氢键:
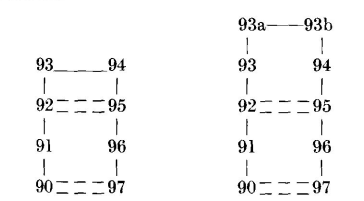
NEWM中残基93和94形成双残基Ⅱ型转角。RHE和KOL中的残基93、93a、93b和94形成了相同构象的四残基转角:它们的主链原子位置的r.m.s.差异为0.19 Å。与KOL和RHE一样,四残基转角几乎在所有转角的第四个位置(即此处的第94位)为Gly或Asn。
根据Kabat等列出的27个人类Vλ结构域和25个小鼠Vλ结构域(L3区序列已知),这些序列中L3区长度的分布情况如下:
六残基的L3区中,预测会像NEWM一样,将91至92和95至96连接到框架β-片层上,而93至94形成一个双残基发夹转角。同样,在具有八个残基的L3区域中,预测91至92和95至96延续β-片层框架,而93、93a、93b和94将形成四残基转角。
REI、MCPC603和J539中的L3区长度相同:
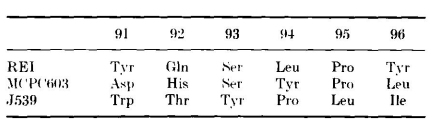
REI和MPCPC603 L3区具有相同的构象:残基91至96的主链原子位置的r.m.s.差异为0.43 Å。而J539中的L3与REI和MCPC603中的L3区具有不同的构象。
通常,对于六残基环,可能会预测残基92和95的主链原子形成氢键,残基93和94形成转角(同Vλ)。而在REI和MCPC603中,这种构象被位于95位的脯氨酸残基所阻止。在这两种Vκ结构域中,残基92具有αL构象,而Pro95具有顺式肽。这使得残基93至96处于伸展构象中(图7)。这种特殊的L3构象的重要决定因素是框架残基90的侧链与其主链原子形成的氢键。虽然90位的侧链不尽相同(REI的侧链是Gln,MCPC603的侧链是Asn),但酰胺的位置相同,作用也相同:NH基团与93和95的羰基形成氢键,O原子与92的酰胺形成氢键(图7)。
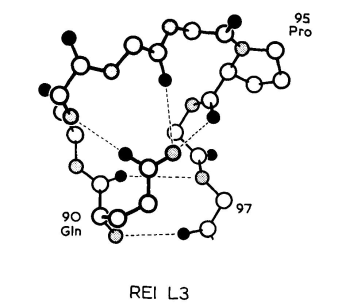
图7 REI Vκ结构域L3区构象
虽然J539中的L3长度为6个残基,但它在95位的残基是Leu而不是Pro,形成了一个两残基的发夹转角:
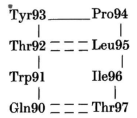
由于在94位有Pro残基,这种转角的构象与Vλ链的不同:Tyr93的ф、φ值为-51°,+131°;Pro94为顺式肽,ф、φ值为-46°,-54°。
Kabat等发现,Vκ序列L3区90和95位的残基是保守的,并认为它们具有结构作用。根据Kabat等列出的121个已知整个L3区序列的人类和小鼠Vκ结构域,L3区域的长度分布如下:
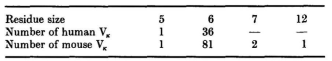
在117个含有6个残基的L3区序列中,93个的95位为Pro,90位为Gln或Asn。它们的长度和序列相同意味着这93个的L3区构象与REI和MCPC603中的相同。还有16个在94位有Pro,但在95位没有,它们很可能具有在J539中发现的L3区构象。
参考文献
Chothia,C. and Lesk,A.M. (1987) Canonical structures for the hypervariable regions of immunoglobulins. J. Mol. Biol., 196, 901–917.

南京德泰生物工程有限公司 Nanjing Detai Bioengineering Co.,Ltd. ©2025 All Rights Reserved